往期回顾
在《可以GPU加速的Bayesian polishing!》一文中,我们介绍了CryoSPARC v4.4发布的Reference-based Motion Correction (RBMC)这一功能,并通过EMPIAR-10025和铁蛋白两套数据集的测试展示了RBMC相对于传统方法的速度优势。文章发出后,读者们对CryoSPARC的新版本和新功能展现了极大热情,同时也对RELION和CryoSPARC两个软件平台间的颗粒转换有了新的需求。因此我们带来新一期的CryoSPARC转RELION流程,希望能够帮助用户利用两个平台的优势充分挖掘数据价值。
本期情境如下
将movie文件导入CryoSPARC,并且使用2D分类进行初步的颗粒筛选。再将颗粒导入RELION中使用2D分类或3D分类对颗粒进一步筛选。之后再将RELION筛选的颗粒导入CryoSPARC中进行Refinement 或RBMC等后续处理。这种情境引入了RELION在2D分类和3D分类上的算法优势,同时充分发挥了CryoSPARC的速度优势和易用性,适用于小蛋白等困难案例的解析。
流程图:
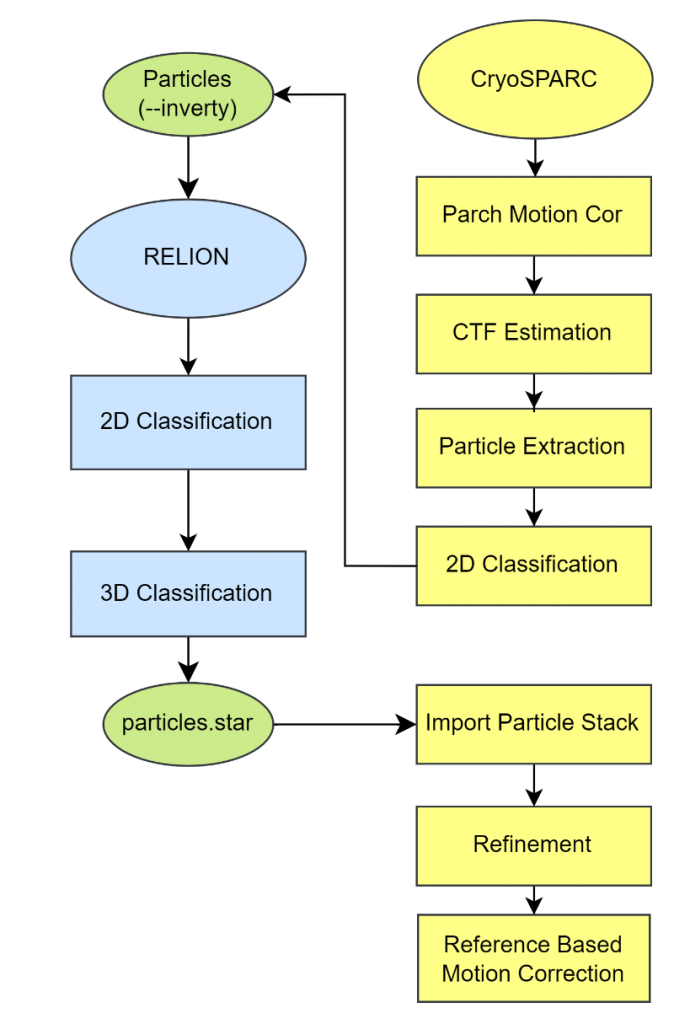
本流程使用的RELION版本为4.0.1,pyem为0.5,CryoSPARC版本为4.4.1。
1. CryoSPARC job情况说明
CryoSPARC项目目录为/ssd/CS-convert
在CryoSPARC中进行Import Movies, Patch Motion Correction, Patch CTF Estimation, Manually Curate Exposures, Blob Picker, Extract From Micrographs, 2D Classification和Select 2D Classes任务
其中,J8为Select 2D Classes任务。备注:此处仅以Select 2D Classes任务作为示例,也可以在CryoSPARC里面进行更多的数据处理之后,再进行转换。
2. CryoSPARC颗粒文件转换
a)将J8中选中的颗粒导出至RELION
进入J8目录,加载pyem环境。对于某些老机器,需要执行最后一步。新机器无需执行最后一步。
/ssd/CS-convert/J8
module load conda
module load pyem
conda activate pyemb)将cs文件转换为star文件,并将star文件拷贝到RELION项目目录下面。注意,此处转换一定要加--inverty参数。
csparc2star.py particles_selected.cs J8_passthrough_particles_selected.cs from_csparc_J8.star --inverty
mkdir /ssd/relion-convert
cp from_csparc_J8.star /ssd/relion-convertc)在RELION目录里对CryoSPARC的颗粒进行软链接
通过head命令查看from_csparc_J8.star文件的前50行,获取颗粒路径
cd /ssd/relion-convert
head -50 from_csparc_J8.star从下图中可以看出,颗粒所在目录的相对路径是J6/extract
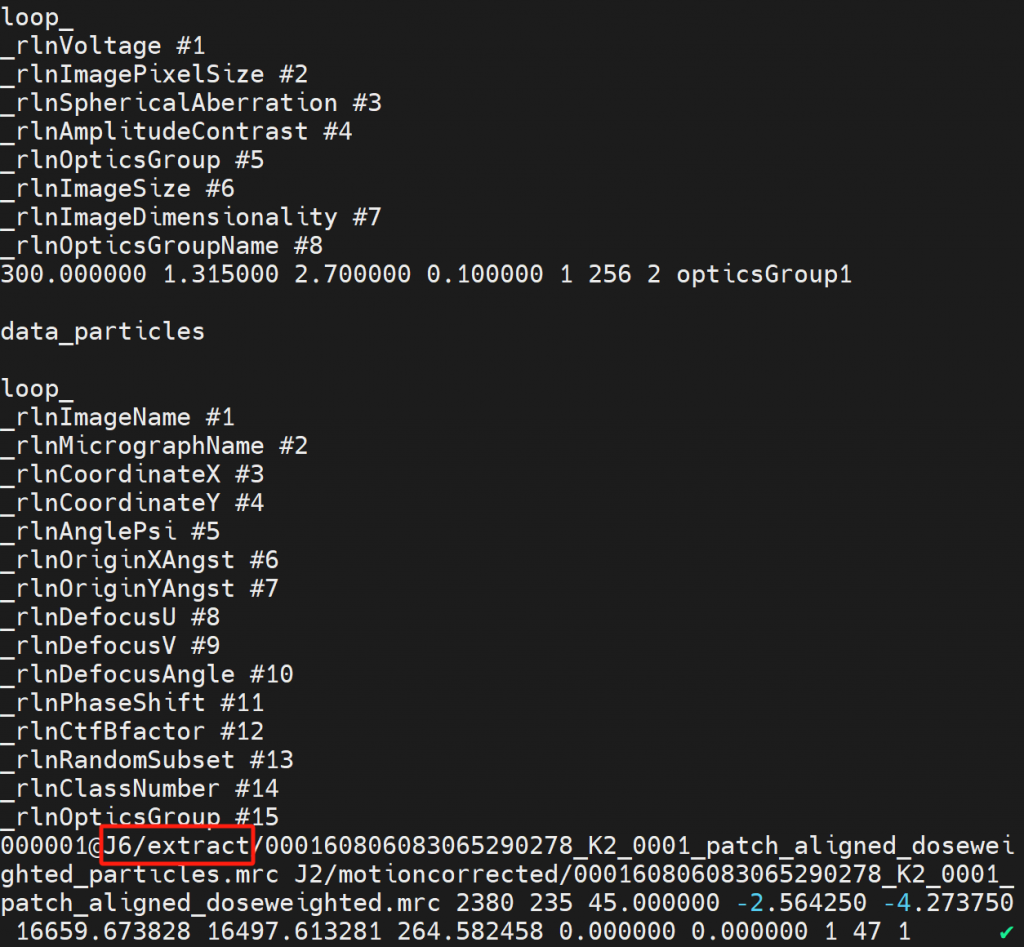
通过执行以下命令,在RELION目录对颗粒文件进行软链接,并对软链接进行重命名。同时将from_csparc_J8.star文件中的particles.mrc字段替换成particles.mrcs字段。
/ssd/relion-convert
mkdir -p J6/extract
ln -s /ssd/CS-convert/J6/extract/*.mrc J6/extract
sed -i 's/particles.mrc/particles.mrcs/g' from_csparc_J8.star
cd /ssd/relion-convert/J6/extract
rename mrc mrcs *.mrc3.RELION数据处理
创建/ssd/relion-convert目录并在目录中启动RELION,然后在Class2D或Class3D任务中输入from_csparc_J8.star文件,并设置相应参数,即可进行二维或三维分类运算,然后使用Subset selection任务进行颗粒筛选,假设该Select任务为job002。
4.CryoSPARC导入颗粒
拷贝Select/job002/particles.star并命名为iny_particles.star,修改star文件中的颗粒后缀名mrcs为mrc,以便在CryoSPARC导入。
cp /ssd/relion-convert/Select/job002/particles.star /ssd/relion-convert/iny_particles.star
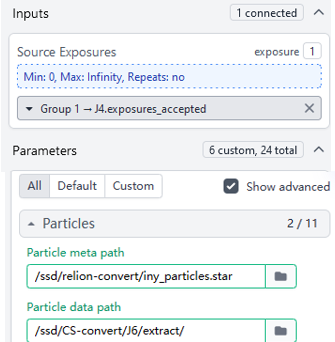
sed -i 's/particles.mrcs/particles.mrc/g' /ssd/relion-convert/iny_particles.star在CryoSPARC创建Import Particle Stack任务,然后将Manually Curate Exposures的结果作为Source Exposures,Particle meta path填入/ssd/relion-convert/iny_particles.star,Particle data path填入/ssd/CS-convert/J6/extract/。点击Queue按钮运行任务。颗粒导入成功后,即可继续进行Refinement和RBMC等任务。
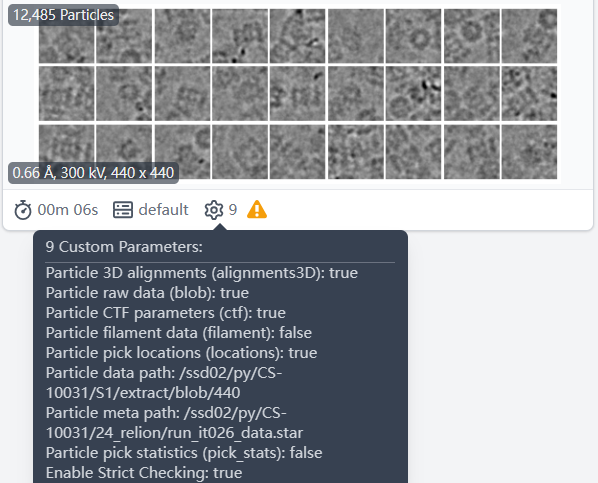
#备注:Import Particle Stack任务中Enable Strict Checking选项关闭时,CryoSPARC将自行判断并提取star文件中的信息,例如:
5. 补充信息
a)为什么转换需要加--inverty参数
当使用CryoSPARC和RELION对同一张图片进行颗粒提取,二者的颗粒坐标有可能存在翻转的情况。在《CryoSparc转Relion进阶流程3》一文中我们介绍过如何通过一张图像判断CryoSPARC和RELION中颗粒坐标的翻转方式,当不确定其转换关系时,我们可以在CryoSPARC中对一张图像进行Import Movies,Patch Motion Correction, Patch CTF Estimation和Manual Picker任务,并挑选几个特征点保存颗粒坐标。
使用pyem的csparc2star.py命令将该任务的颗粒文件进行转换:
假设该任务的路径为/ssd/CS-convert/J10。
cd /ssd/CS-convert/J10
csparc2star.py extracted_particles.cs from_csparc_J10.star 在CryoSPARC中创建Import Particle Stack任务,并导入from_csparc_J10.star。然后使用Extract From Micrographs任务重新提取颗粒。该任务会输出颗粒在图像上的位置。

对比两张图片可以看出,转换后的颗粒和原坐标存在沿y轴的翻转,因此使用csparc2star.py命令时需要添加--inverty。读者还可据此对比--invertx,--inverty或参数组合使用的不同效果。
#注意 在CryoSPARC和pyem更新后,默认参数可能有所改变,因此需要根据该方法重新找寻规律。
b)不加--inverty参数的影响
如果不加--inverty参数,不会影响RELION中的数据处理结果。另外,如果在CryoSPARC的Extract From Micrographs任务里开启了Flip mic. in y before extract?,也可以正确地提取颗粒。
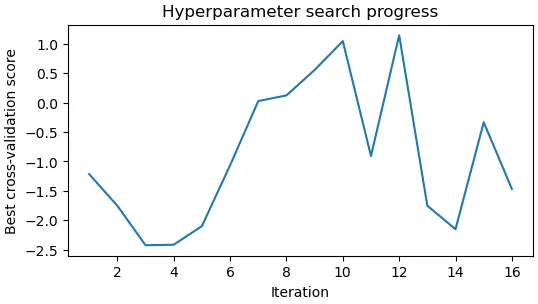
但是上述操作是让micrographs在提取颗粒时进行了上下翻转,而非让颗粒坐标进行了上下翻转。因此在运行RBMC任务时,它使用的颗粒坐标依然不正确,这将导致计算超参数hyperparameters速度变慢,并影响Hyperparameters search progress和empirical dose weights的结果(下图)。
以上就是CryoSPARC转RELION进阶流程4的全部内容了,下期我们将带来新情境:RELION>-Refine3D>-CryoSPARC>-RBMC的转换,各位读者下期见!
